Экзон

Экзон — гэта любая частка гена, якая будзе ўтвараць частку канчатковай РНК, пасля выдалення інтронаў шляхам сплайсінгу РНК. Тэрмін экзон адносіцца як да паслядоўнасці ДНК у гене, так і да адпаведнай паслядоўнасці ў транскрыптах РНК. Пры сплайсінгу РНК інтроны выдаляюцца, а экзоны кавалентна злучаюцца адзін з адным у рамках генерацыі спелай РНК. Гэтак жа, як увесь набор генаў віду складае геном, увесь набор экзонаў складае экзом.
Гісторыя[правіць | правіць зыходнік]
Тэрмін экзон паходзіць ад экспрэсіраванай вобласці (англ. expressed region) і быў уведзены амерыканскім біяхімікам Уолтэрам Гілбертам у 1978 годзе: «Паняцце цыстрона … павінна быць заменена на паняцце адзінка транскрыпцыі, якая змяшчае вобласці, якія будуць страчаны ад спелага месэнджара — якія я прапаную называць інтронамі (для ўнутрыгенных рэгіёнаў), што чаргуюцца з рэгіёнамі, якія будуць экспрэсіравацца — экзоны».[1]
Гэта вызначэнне першапачаткова было зроблена для бялок-кадуючых транскрыптаў, якія злучаюцца перад трансляцыяй. Пазней гэты тэрмін стаў уключаць паслядоўнасці, выдаленыя з рРНК[2] і тРНК,[3] і іншых нкРНК,[4] а таксама пазней ён выкарыстоўваўся для малекул РНК, якія транскрыбіруюцца з розных частак геному, а затым лігіруюць шляхам транс-сплайсінгу.[5]
Уклад у геномы і размеркаванне па памерах[правіць | правіць зыходнік]
Хаця аднаклетачныя эўкарыёты, такія як дрожджы, альбо не маюць інтронаў, альбо іх вельмі мала, метазоі і асабліва геномы пазваночных маюць вялікую долю некадуючай ДНК. Напрыклад, у геноме чалавека толькі 1,1 % геному займаюць экзоны, тады як 24 % з’яўляецца інтронамі, прычым 75 % геному складае міжгенная ДНК.[6] Гэта можа даць практычныя перавагі ў ахове здароўя (напрыклад, у персаналізаванай медыцыне), таму што гэта робіць камерцыялізаванае секвенаванне цэлага экзома меншай і менш дарагой задачай, чым камерцыялізаванае секвенаванне цэлага геному. Вялікая варыяцыя памеру геному і С-значэння для розных формаў жыцця паставіла цікавую задачу, якая называецца загадкай C-значэння.
Ва ўсіх эўкарыётычных генах у GenBank было (у 2002 г.) у сярэднім 5,48 экзона на ген, які кадуе бялок. У сярэднім адзін экзон кадуе 30-36 амінакіслот.[7] У той час як самы доўгі экзон у геноме чалавека мае даўжыню 11555 пар нуклеатыдаў, было выяўлена, што некаторыя экзоны маюць даўжыню ўсяго 2 п.н., чаго не хапае для кадзіроўкі нават 1 амінакіслаты.[8] Аднануклеатыдны экзон быў знойдзены ў геноме Arabidopsis.[9] У людзей, як мРНК, якая кадуе бялок, так і большасць некадуючых РНК таксама маюць некалькі экзонаў.[10]
Будова і функцыі[правіць | правіць зыходнік]
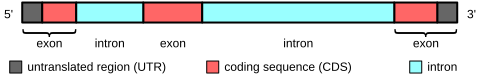
У генах, якія кадуюць бялок, экзоны ўключаюць як паслядоўнасць, якая кадуе бялок, так і 5'- і 3'- нетранслюемыя вобласці (UTR, ад англ. untranslated region). Часта першы экзон мае як 5'-UTR, так і першую частку кадуючай паслядоўнасці, але экзоны, якія змяшчаюць толькі ўчасткі 5'-UTR або (радзей) 3'-UTR, сустракаюцца ў некаторых генах, гэта значыць UTR могуць утрымліваць інтроны.[11] Некаторыя некадуючыя транскрыпты РНК таксама маюць экзоны і інтроны.
Спелыя мРНК, якія паходзяць з аднаго гена, не павінны ўключаць аднолькавыя экзоны, паколькі розныя экзоны ў прэ-мРНК могуць быць выдалены ў працэсе альтэрнатыўнага сплайсінгу.
Экзанізацыя — гэта стварэнне новага экзона ў выніку мутацый у паслядоўнасці інтронаў.[12]
Эксперыментальныя падыходы з выкарыстаннем экзонаў[правіць | правіць зыходнік]
Захоп экзонаў або «захоп генаў» — гэта метад малекулярнай біялогіі, які выкарыстоўвае існаванне сплайсінгу для пошуку новых генаў.[13] Першы экзон «захопленага» гена зрошчваецца з экзонам, які змяшчаецца ва ўстаўной ДНК. Гэты новы экзон змяшчае адкрытую рамку счытвання (ORF) для гена-рэпарцёра, які зараз можа быць экспрэсаваны з дапамогай энхансераў, якія кантралююць мэтавы ген. Навуковец ведае, што пры экспрэсіі рэпарцёрнага гена быў захоплены новы ген.
Сплайсінг можа быць эксперыментальна мадыфікаваны такім чынам, каб мэтавыя экзоны былі выключаны з фінальных транскрыптаў мРНК шляхам блакіроўкі доступу малых ядзерных рыбануклеапратэінавых часціц (snRNP), якія накіроўваюць сплайсінг, да прэ-мРНК з дапамогай марфалінавых алігануклеатыдаў.[14] Гэта стала стандартнай методыкай у біялогіі развіцця. Марфалінавыя алігамеры таксама можна нацэліць на прадухіленне малекул, якія рэгулююць сплайсінг (напрыклад, узмацняльнікі сплайсінгу, супрэсары сплайсінгу), ад звязвання з прэ-мРНК, змяняючы патэрны сплайсінгу.
Няправільнае выкарыстанне тэрміна[правіць | правіць зыходнік]
Распаўсюджана няправільнае выкарыстанне тэрміна экзон: «экзоны кадуюць бялок», «экзоны кадуюць амінакіслоты» або «экзоны транслююцца». Аднак такія азначэнні ахопліваюць толькі гены, якія кадуюць бялкі, і прапускаюць тыя экзоны, якія становяцца часткай некадуючай РНК[15] або нетранслюемай вобласці мРНК.[16][17] Такія няслушныя азначэнні ўсё яшчэ сустракаюцца ў аўтарытэтных другасных крыніцах.[18][19]
Гл. таксама[правіць | правіць зыходнік]
Крыніцы[правіць | правіць зыходнік]
- ↑ Gilbert W (February 1978). "Why genes in pieces?". Nature. 271 (5645): 501. Bibcode:1978Natur.271..501G. doi:10.1038/271501a0. PMID 622185.
- ↑ Kister KP, Eckert WA (March 1987). "Characterization of an authentic intermediate in the self-splicing process of ribosomal precursor RNA in macronuclei of Tetrahymena thermophila". Nucleic Acids Research. 15 (5): 1905–20. doi:10.1093/nar/15.5.1905. PMC 340607. PMID 3645543.
- ↑ Valenzuela P, Venegas A, Weinberg F, Bishop R, Rutter WJ (January 1978). "Structure of yeast phenylalanine-tRNA genes: an intervening DNA segment within the region coding for the tRNA". Proceedings of the National Academy of Sciences of the United States of America. 75 (1): 190–4. Bibcode:1978PNAS...75..190V. doi:10.1073/pnas.75.1.190. PMC 411211. PMID 343104.
- ↑ Khan, MR; Wellinger, RJ; Laurent, B (August 2021). "Exploring the Alternative Splicing of Long Noncoding RNAs". Trends in Genetics. 37 (8): 695–698. doi:10.1016/j.tig.2021.03.010. PMID 33892960. S2CID 233382870.
- ↑ Liu AY, Van der Ploeg LH, Rijsewijk FA, Borst P (June 1983). "The transposition unit of variant surface glycoprotein gene 118 of Trypanosoma brucei. Presence of repeated elements at its border and absence of promoter-associated sequences". Journal of Molecular Biology. 167 (1): 57–75. doi:10.1016/S0022-2836(83)80034-5. PMID 6306255.
- ↑ Venter J.C.; et al. (2000). "The Sequence of the Human Genome". Science. 291 (5507): 1304–51. Bibcode:2001Sci...291.1304V. doi:10.1126/science.1058040. PMID 11181995.
- ↑ Sakharkar M, Passetti F, de Souza JE, Long M, de Souza SJ (2002). "ExInt: an Exon Intron Database". Nucleic Acids Res. 30 (1): 191–4. doi:10.1093/nar/30.1.191. PMC 99089. PMID 11752290.
- ↑ Sakharkar M.K.; Chow VT; Kangueane P. (2004). "Distributions of exons and introns in the human genome". In Silico Biol. 4 (4): 387–93. PMID 15217358.
- ↑ Guo Lei, Liu Chun-Ming (2015). "A single-nucleotide exon found in Arabidopsis". Scientific Reports. 5: 18087. Bibcode:2015NatSR...518087G. doi:10.1038/srep18087. PMC 4674806. PMID 26657562.
- ↑ Derrien, T; Johnson, R; Bussotti, G; Tanzer, A; Djebali, S; Tilgner, H; Guernec, G; Martin, D; Merkel, A; Knowles, DG; Lagarde, J; Veeravalli, L; Ruan, X; Ruan, Y; Lassmann, T; Carninci, P; Brown, JB; Lipovich, L; Gonzalez, JM; Thomas, M; Davis, CA; Shiekhattar, R; Gingeras, TR; Hubbard, TJ; Notredame, C; Harrow, J; Guigó, R (September 2012). "The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression". Genome Research. 22 (9): 1775–89. doi:10.1101/gr.132159.111. PMC 3431493. PMID 22955988.
- ↑ Bicknell, AA (December 2012). "Introns in UTRs: Why we should stop ignoring them". BioEssays. 34 (12): 1025–1034. doi:10.1002/bies.201200073. PMID 23108796. S2CID 5808466.
- ↑ Sorek R (October 2007). "The birth of new exons: mechanisms and evolutionary consequences". RNA. 13 (10): 1603–8. doi:10.1261/rna.682507. PMC 1986822. PMID 17709368.
- ↑ Duyk G. M; Kim S. W.; Myers R. M; Cox D. R (1990). "Exon Trapping: a Genetic Screen to Identify Candidate Transcribed Sequences in Cloned Mammalian Genomic DNA". Proceedings of the National Academy of Sciences. 87 (22): 8995–8999. Bibcode:1990PNAS...87.8995D. doi:10.1073/pnas.87.22.8995. PMC 55087. PMID 2247475.
- ↑ Morcos PA (June 2007). "Achieving targeted and quantifiable alteration of mRNA splicing with Morpholino oligos". Biochemical and Biophysical Research Communications. 358 (2): 521–7. doi:10.1016/j.bbrc.2007.04.172. PMID 17493584.
- ↑ Khan, MR; Wellinger, RJ; Laurent, B (August 2021). "Exploring the Alternative Splicing of Long Noncoding RNAs". Trends in Genetics. 37 (8): 695–698. doi:10.1016/j.tig.2021.03.010. PMID 33892960. S2CID 233382870.
- ↑ Lu, J; Williams, JA; Luke, J; Zhang, F; Chu, K; Kay, MA (January 2017). "A 5' Noncoding Exon Containing Engineered Intron Enhances Transgene Expression from Recombinant AAV Vectors in vivo". Human Gene Therapy. 28 (1): 125–134. doi:10.1089/hum.2016.140. PMC 5278795. PMID 27903072.
- ↑ Chung, BY; Simons, C; Firth, AE; Brown, CM; Hellens, RP (19 May 2006). "Effect of 5'UTR introns on gene expression in Arabidopsis thaliana". BMC Genomics. 7: 120. doi:10.1186/1471-2164-7-120. PMC 1482700. PMID 16712733.
- ↑ Exon (англ.). Genome.gov. Архівавана з першакрыніцы 16 сакавіка 2023. Праверана 23 сакавіка 2023.
- ↑ Exon (англ.). www.nature.com. Scitable. Архівавана з першакрыніцы 23 сакавіка 2023. Праверана 23 сакавіка 2023.
Бібліяграфія[правіць | правіць зыходнік]
- Zhang MQ (May 1998). "Statistical features of human exons and their flanking regions". Human Molecular Genetics. 7 (5): 919–32. doi:10.1093/hmg/7.5.919. PMID 9536098.
- Thanaraj TA, Robinson AJ (November 2000). "Prediction of exact boundaries of exons". Brief. Bioinform. 1 (4): 343–56. doi:10.1093/bib/1.4.343. PMID 11465052.
