Інтрон
Інтрон — гэта паслядоўнасць нуклеатыдаў у гене, якая не экспрэсуецца і не прысутная ў канчатковым прадукце РНК. Слова інтрон паходзіць ад тэрміна intragenic region, то-бок вобласць унутры гена.[1] Тэрмін інтрон адносіцца як да паслядоўнасці ДНК у гене, так і да адпаведнай паслядоўнасці РНК у транскрыптах РНК.[2] Неінтронныя паслядоўнасці, якія злучаюцца падчас працэсінгу РНК з фармаваннем спелай РНК, называюцца экзонамі.[3]
Інтроны знаходзяцца ў генах большасці арганізмаў і многіх вірусаў, і яны могуць знаходзіцца як у генах, што кадуюць бялок, так і ў генах, якія функцыянуюць як РНК (некадуючыя гены). Існуе чатыры асноўных тыпа інтронаў: інтроны тРНК, інтроны групы I, інтроны групы II і сплайсасомныя інтроны (гл. ніжэй). Інтроны рэдка сустракаюцца ў бактэрый і архей (пракарыёт), але большасць эўкарыятычных генаў змяшчае некалькі сплайсасомных інтронаў.
Адкрыццё і этымалогія
[правіць | правіць зыходнік]Упершыню інтроны былі выяўлены ў бялок-кадуючых генах адэнавірусу[4][5], а потым ідэнтыфікаваныя ў генах транспартнай і рыбасомнай РНК. Зараз вядома, што інтроны сустракаюцца ў самых розных генах арганізмаў, бактэрый[6] і вірусаў ва ўсіх біялагічных царствах.
Той факт, што гены расшчапляюцца або перарываюцца інтронамі, быў адкрыты незалежна адзін ад аднаго ў 1977 годзе Філіпам Аленам Шарпам і Рычардам Дж. Робертсам, за што яны разам атрымалі Нобелеўскую прэмію па фізіялогіі і медыцыне ў 1993 годзе[7]. Тэрмін інтрон быў уведзены амерыканскім біяхімікам Уолтэрам Гілбертам:[1]
«Паняцце цыстрона [г.зн. гена] … павінна быць заменена на паняцце адзінкі транскрыпцыі, якая змяшчае вобласці, што будуць страчаны ад спелага мэсэнджара — іх я прапаную называць інтронамі (для ўнутрыгенных абласцей) — якія чаргуюцца з абласцямі, што будуць экспрэсаваныя — экзонамі». (Гілберт 1978)
Тэрмін інтрон таксама адносіцца да інтрацыстрона, то-бок дадатковай часткі ДНК, якая ўзнікае ў цыстроне.[8]
Хоць інтроны часам называюць прамежкавымі паслядоўнасцямі,[9] тэрмін «прамежкавая паслядоўнасць» можа адносіцца да любога з некалькіх сямействаў унутраных паслядоўнасцей нуклеінавых кіслот, якія не прысутнічаюць у канчатковым прадукце гена, уключаючы інтэіны, нетранслюемыя вобласці (UTR) і нуклеатыды, што выдаляюцца шляхам рэдагавання РНК.
Размеркаванне
[правіць | правіць зыходнік]Назіраецца, што частата інтронаў у розных геномах моцна адрозніваецца сярод біялагічных арганізмаў. Напрыклад, інтроны надзвычай часта сустракаюцца ў ядзерным геноме пазваночных з сківіцай (напрыклад, чалавека, мышэй і рыб-фугу), дзе кадуючыя бялок гены амаль заўсёды маюць некалькі інтронаў, у той час як інтроны рэдка сустракаюцца ў ядзерных генах некаторых эўкарыятычных мікраарганізмаў,[10] напрыклад, у пякарскіх/піўных дражджах (Saccharomyces cerevisiae). Мітахандрыяльныя геномы пазваночных увогуле не маюць інтронаў.[11]
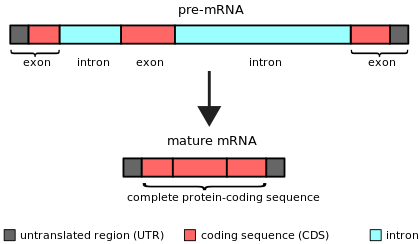
Асабліва экстрэмальны выпадак — ген dhc7 дразафілы, які змяшчае інтрон памерам ≥3,6 мегабазы (Mb), транскрыпцыя якога займае прыкладна тры дні.[12][13] З іншага боку, даследаванне 2015 г. паказвае, што самая кароткая вядомая даўжыня інтрона ў жывёл складае 30 пар асноў (п.н.), якія знаходзяцца ў гене MST1L чалавека.[14] Самыя кароткія вядомыя інтроны належаць рознарэснічным інфузорыям, такім як Stentor coeruleus, у якіх большасць (> 95 %) інтронаў даўжынёй 15 або 16 п.н.[15]
Класіфікацыя
[правіць | правіць зыходнік]Сплайсінг усіх інтронзмяшчальных малекул РНК вонкава падобны. Тым не менш, розныя тыпы інтронаў былі ідэнтыфікаваныя шляхам вывучэння структуры інтронаў з дапамогай аналізу паслядоўнасці ДНК разам з генетычным і біяхімічным аналізам рэакцый сплайсінгу РНК. Вылучана як мінімум чатыры розныя класы інтронаў:
- Інтроны ў ядзерных кадуючых бялок генах, якія выдаляюцца сплайсасомамі (сплайсасомныя інтроны)
- Інтроны ў ядзерных і архейных генах транспартнай РНК, якія выдаляюцца бялкамі (інтроны тРНК)
- Інтроны групы I, якія выдаляюцца самастойна РНК-каталізам
- Інтроны групы II, якім характэрны аўтасплайсінг, як і інтронам I групы.
Мяркуецца, што інтроны III групы з’яўляюцца пятым класам, але мала што вядома пра біяхімічны апарат, які забяспечвае іх сплайсінг. Іх часта адносяць да II групы праз структурнае і функцыянальнае падабенства. Інтроны I, II, III груп самастойна выразаюцца з мРНК і менш распаўсюджаныя, чым сплайсасомныя інтроны.[16]
Сплайсасомныя інтроны
[правіць | правіць зыходнік]Ядзерныя інтроны ў прэ-мРНК (сплайсасомныя інтроны) характарызуюцца спецыфічнымі паслядоўнасцямі, размешчанымі на межах паміж інтронамі і экзонамі.[17] Гэтыя паслядоўнасці распазнаюцца малекуламі сплайсасомнай РНК, калі ініцыююцца рэакцыі сплайсінгу.[18] Акрамя таго, яны маюць кропку разгалінавання, пэўную паслядоўнасць нуклеатыдаў паблізу 3'-канца інтрона, якая кавалентна злучаецца з 5'-канцом інтрона ў працэсе сплайсінгу, утвараючы разгалінаваны інтрон. Акрамя гэтых трох кароткіх кансерватыўных элементаў, паслядоўнасці інтронаў ядзернай прэ-мРНК вельмі зменлівыя. Ядзерныя інтроны прэ-мРНК часта значна даўжэйшыя за суседнія экзоны.
Выдаленне інтронаў транспартнай РНК залежыць ад бялкоў. Выразанне адбываецца ў пэўным месцы ўнутры антыкадонавай пятлі несплайсаваных папярэднікаў тРНК і выдаляюцца інтроны эндануклеазай сплайсінгу тРНК. Затым экзоны злучаюцца разам другім бялком, тРНК-лігазай сплайсінгу.[19] Інтроны, якім характерны самасплайсінг, таксама часам сустракаюцца ў генах тРНК.[20]
Інтроны I і II групы
[правіць | правіць зыходнік]Інтроны групы I і групы II знаходзяцца ў генах, якія кадуюць бялкі (інфармацыйная РНК), транспартную РНК і рыбасамальную РНК у вельмі шырокім дыяпазоне жывых арганізмаў.[21][22] Пасля транскрыпцыі ў РНК інтроны груп I і II таксама ўступаюць у шырокія ўнутраныя ўзаемадзеянні, якія дазваляюць ім згортвацца ў спецыфічную складаную трохмерную архітэктуру. Гэтыя складаныя архітэктуры дазваляюць некаторым інтронам групы I і групы II самастойна выдаляцца, гэта значыць малекула РНК, якая змяшчае інтрон, можа перабудоўваць сваю ўласную кавалентную структуру так, каб дакладна выдаліць інтрон і злучыць экзоны разам у правільным парадку. У некаторых выпадках пэўныя бялкі, якія звязваюць інтроны, удзельнічаюць у сплайсінгу, дапамагаючы інтрону згортвацца ў трохмерную структуру, неабходную для аўтасплайсінгу. Інтроны групы I і групы II адрозніваюцца рознымі наборамі ўнутраных кансерватыўных паслядоўнасцей і трохмерных структур. Таксама падчас сплайсінгу малекул РНК, што маюць інтроны групы II, ствараюцца разгалінаваныя інтроны (як і інтроны сплайсасомных РНК), у той час як інтроны групы I выкарыстоўваюць некадуемы гуаназінавы нуклеатыд (звычайна GTP), каб ініцыяваць сплайсінг, дадаючы яго да 5'-канца выдаленага інтрона.
Дакладнасць сплайсінгу
[правіць | правіць зыходнік]Сплайсасома — гэта вельмі складаная структура, у якую ўваходзіць да ста бялкоў і пяць розных РНК. Субстратам рэакцыі з’яўляецца доўгая малекула РНК, і рэакцыі трансэтэрыфікацыі, якія каталізуюцца сплайсасомай, патрабуюць аб’яднання сайтаў, якія могуць знаходзіцца на тысячах нуклеатыдаў адзін ад аднаго.[23][24] Усе біяхімічныя рэакцыі звязаны з вядомай частатой памылак, і чым больш складаная рэакцыя, тым вышэй верагоднасць памылкі. Такім чынам, не дзіўна, што рэакцыя сплайсінгу, каталізуемая сплайсасомай, мае значную долю памылак, нават калі існуюць дадатковыя фактары сплайсасомы, якія падаюляўць выпадковае расшчапленне скрытых сайтаў сплайсінгу.[25]
У ідэальных умовах рэакцыя сплайсінгу, верагодна, будзе дакладнай на 99,999 % (частата памылак 10−5), правільныя экзоны будуць аб’яднаны, а правільны інтрон будзе выдалены.[26] Аднак гэтыя ідэальныя ўмовы патрабуюць вельмі блізкага супадзення з найлепшымі паслядоўнасцямі сайтаў сплайсінгу і адсутнасці якіх-небудзь канкуруючых скрытых паслядоўнасцей сайтаў сплайсінгу ў інтронах, і гэтыя ўмовы рэдка сустракаюцца ў вялікіх эукарыятычных генах, якія могуць ахопліваць больш за 40 тысяч пар асноў. Нядаўнія даследаванні паказалі, што фактычны ўзровень памылак можа быць значна вышэйшым за 10−5 і дасягаць 2 % або 3 % памылак (частата памылак 2 або 3 x 10 −2) на ген.[27][28][29] Дадатковыя даследаванні паказваюць, што доля памылак складае не менш за 0,1 % на інтрон.[30][31] Гэты адносна высокі ўзровень памылак сплайсінгу тлумачыць, чаму большасць варыянтаў сплайсінгу хутка разбураюцца нонсэнс-апасродкаваным распадам.[32][33]
Наяўнасць неакуратных сайтаў звязвання ў генах выклікае памылкі сплайсінгу, і можа здацца дзіўным, што гэтыя сайты не былі ліквідаваны натуральным адборам. Аргумент аб іх устойлівасці падобны на аргумент аб прычыне існавання «смеццевай» ДНК.[30][34]
Хаця мутацыі, якія ствараюць або разбураюць сайты звязвання, могуць быць крыху шкоднымі, вялікая колькасць магчымых такіх мутацый робіць непазбежным тое, што некаторыя з іх дасягнуць фіксацыі ў папуляцыі. Гэта асабліва актуальна для такіх відаў, як людзі, з адносна невялікімі доўгатэрміновымі эфектыўнымі памерамі папуляцыі. Такім чынам, праўдападобна, што геном чалавека нясе значную нагрузку субаптымальных паслядоўнасцей, якія выклікаюць генерацыю аберантных ізаформ транскрыптаў.[30]
У той час як каталітычная рэакцыя можа быць дастаткова дакладнай для эфектыўнага працэсінгу большую частку часу, агульны ўзровень памылак можа быць часткова абмежаваны дакладнасцю транскрыпцыі, таму што памылкі транскрыпцыі прывядуць да мутацыі, якія ствараюць скрытыя сайты сплайсінгу. Акрамя таго, частата памылак транскрыпцыі 10−5 — 10−6 дастаткова высокая, каб адзін з кожных 25 000 транскрыбаваных экзонаў меў памылку ўключэння ў адным з сайтаў сплайсінгу, што вядзе да прапушчанага інтрона або экзона. Амаль усе гены з вялікай колькасцю экзонаў будуць ствараць няправільна сплайсаваныя транскрыпты, але частата гэтага фонавага шуму будзе залежаць ад памеру генаў, колькасці інтронаў і якасці паслядоўнасцей сайтаў сплайсінгу.[28][31]
У некаторых выпадках варыянты сплайсінгу ўтвараюцца ў выніку мутацый у гене. Гэта могуць быць SNP-палімарфізмы, якія ствараюць схаваны сайт сплайсінгу або муціруюць функцыянальны сайт. Таксама могуць адбывацца мутацыі саматычных клетак, якія ўплываюць на сплайсінг ў пэўнай тканцы або цэлявай лініі.[35][36][37] Калі мутантны алель знаходзіцца ў гетэразіготным стане, гэта прыводзіць да ўтварэння двух распаўсюджаных варыянтаў сплайсінгу: аднаго функцыянальнага і аднаго нефункцыянальнага. У гомазіготным стане мутантныя алелі могуць выклікаць генетычныя захворванні, такія як гемафілія, якая выявілася ў нашчадкаў каралевы Вікторыі, калі мутацыя ў адным з інтронаў гена фактару згортвання крыві стварае схаваны 3'-сайт сплайсінгу, які прыводзіць да аберантнага сплайсінгу.[38] Значная частка смерцяў людзей ад хвароб можа быць выклікана мутацыямі, што парушаюць нармальны сплайсінг; галоўным чынам, шляхам стварэння схаваных сайтаў сплайсінгу.[39][36]
Няправільна сплайсаваныя транскрыпты могуць быць лёгка выяўленыя, і іх паслядоўнасці могуць быць занесены ў анлайн базы даных. Звычайна іх апісваюць як «альтэрнатыўна сплайсаваныя» транскрыпты, што можа прывесці да блытаніны, паколькі гэты тэрмін не праводзіць адрозненні паміж рэальным, біялагічна значным альтэрнатыўным сплайсінгам і шумам апрацоўкі праз памылкі сплайсінгу. Адным з цэнтральных пытанняў у галіне альтэрнатыўнага сплайсінгу з’яўляецца высвятленне адрозненняў паміж гэтымі двума працэсамі. Многія навукоўцы сцвярджалі, што нулявая гіпотэза павінна быць сплайсінгавым шумам, ускладаючы цяжар даказвання на тых, хто сцвярджае аб біялагічна значным альтэрнатыўным сплайсінгу. На думку гэтых навукоўцаў, сцвярджэнне аб функцыянальнасці павінна суправаджацца пераканаўчымі доказамі таго, што з аднаго і таго ж гена кадуецца мноства функцыянальных прадуктаў.[40][41]
Біялагічныя функцыі і эвалюцыя
[правіць | правіць зыходнік]Хоць інтроны і не кадуюць бялковыя прадукты, яны з’яўляюцца неад’емнай часткай рэгуляцыі экспрэсіі генаў. Некаторыя інтроны самі кадуюць функцыянальныя РНК з дапамогай далейшай апрацоўкі пасля сплайсінгу з утварэннем некадуючых малекул РНК.[42] Альтэрнатыўны сплайсінг шырока выкарыстоўваецца для атрымання мноства бялкоў з аднаго гена. Акрамя таго, некаторыя інтроны маюць важную ролю ў шырокім спектры рэгуляторных функцый да экспрэсіі генаў, напрыклад, такіх як нонсэнс-апасродкаваны распад[43] і экспарт мРНК[44].
Пасля першапачатковага адкрыцця інтронаў у ядзерных генах эукарыёт, што кадуюць бялок, існавалі сур’ёзныя дэбаты наконт таго, ці былі інтроны ў сучасных арганізмах унаследаваны ад агульнага продка (так званая гіпотэза ранніх інтронаў), ці яны з’явіліся ў генах адносна нядаўна ў эвалюцыйным працэсе (гіпотэза позніх інтронаў). Іншая тэорыя заключаецца ў тым, што сплайсасома і інтрон-экзонная структура генаў з’яўляюцца рэліктам свету РНК.[45] Дагэтуль вядуцца сур’ёзныя спрэчкі наконт таго, наколькі правільная з гэтых гіпотэз, але на гэты момант папулярны кансенсус заключаецца ў тым, што пасля ўтварэння першай эўкарыятычнай клеткі інтроны групы II з бактэрыяльнага эндасімбіёнта ўварваліся ў геном гаспадара. Напачатку гэтыя самасплайсуючыяся інтроны выдаляліся з прэ-мРНК, але з цягам часу некаторыя з іх страцілі гэтую здольнасць, і іх выдаленню прыйшлося дапамагаць транс-рэгуляторнымі механізмамі з дапамогай іншых інтронаў групы II. У рэшце рэшт развіўся шэраг спецыфічных трансактыўных інтронаў, якія сталі папярэднікамі snRNAs сплайсасомы. Эфектыўнасць сплайсінгу была палепшана шляхам асацыяцыі з стабілізуючымі бялкамі з фармаваннем прымітыўнай сплайсасомы.[46][47][48][49]
Раннія даследаванні паслядоўнасцей геномнай ДНК з шырокага спектру арганізмаў паказваюць, што інтрон-экзонная структура гамалагічных генаў у розных арганізмах можа моцна адрознівацца.[50] Больш нядаўнія даследаванні цэлых геномаў эўкарыётаў паказалі, што даўжыня і шчыльнасць (інтроны/ген) інтронаў істотна вар’іруецца ў розных роднасных відаў. Напрыклад, калі геном чалавека змяшчае ў сярэднім 8,4 інтронаў на ген (139 418 у геноме), аднаклетачны грыб Encephalitozoon cuniculi мае толькі 0,0075 інтронаў на 1 ген (15 інтронаў на ўвесь геном).[51] Паколькі эўкарыёты ўзніклі ад агульнага продка, падчас эвалюцыі павінна было адбыцца значнае распаюсюджанне або страта інтронаў.[52][53] Лічыцца, што гэты працэс падвяргаецца адбору з тэндэнцыяй да павелічэння колькасці інтронаў у больш буйных відаў з-за меншага эфектыўнага памеру папуляцыі, і наадварот у меншых (асабліва аднаклетачных) відаў.[54] Біялагічныя фактары таксама ўплываюць на тое, якія гены ў геноме губляюць або назапашваюць інтроны.[55][56][57]
Альтэрнатыўны сплайсінг экзонаў у гене пасля выдалення інтронаў спрыяе ўвядзенню большай варыябельнасці бялковых паслядоўнасцей, трансляваных з аднаго гена, што дазваляе генераваць некалькі роднасных бялкоў з аднаго гена і аднаго мРНК транскрыпту. Кантроль альтэрнатыўнага РНК сплайсінгу ажыццяўляецца складанай сеткай сігнальных малекул, якія рэагуюць на шырокі спектр ўнутрыцэлявых і пазацэлявых сігналаў.
Вядома, што некаторыя інтроны ўзмацняюць экспрэсію гена, у якім яны знаходзяцца, з дапамогай працэсу, які называюць інтрон-апасродкаваным узмацненнем (IME).
Актыўна транскрыбуемыя вобласці ДНК часта ўтвараюць R-петлі, якія ўразлівыя да пашкоджання ДНК. У высокаэкспрэсуемых генах дрожджаў інтроны інгібіруюць утварэнне R-пяцель і ўзнікненне пашкоджанняў ДНК.[58] Агульнагеномны аналіз як у дрожджаў, так і ў чалавека паказаў, што гены, якія змяшчаюць інтроны, маюць меншую распаўсюджанасць R-пяцель і памяншаюць пашкоджанні ДНК у параўнанні з генамі, якія не маюць інтронаў і валодаюць падобнай экспрэсіяй.[58] Устаўка інтрона ў ген, схільнага да ўтварэння R-пяцель, таксама можа падаўляць утварэнне і рэкамбінацыю R-пяцель. Боне і яго калегі[58] выказалі здагадку, што функцыя інтронаў у падтрыманні генетычнай стабільнасці можа тлумачыць іх эвалюцыйнае захаванне ў пэўных месцах, асабліва ў генах з высокай экспрэсіяй.
Адаптацыя да галадання
[правіць | правіць зыходнік]Фізічная прысутнасць інтронаў спрыяе ўстойлівасці цэляў да галадання праз узмоцненую інтронамі рэпрэсію генаў рыбасамальных бялкоў, што прымаюць удзел у шляхах успрымання пажыўных рэчываў.[59]
Як мабільныя генетычныя элементы
[правіць | правіць зыходнік]Інтроны могуць быць страчаны або атрыманы на працягу эвалюцыйнага шляху, як паказана ў шматлікіх параўнальных даследаваннях арталагічных генаў. Наступны аналіз выявіў тысячы прыкладаў страты і павелічэння колькасці інтронаў, і была выказана здагадка, што з’яўленне эўкарыёт, або пачатковыя этапы эвалюцыі эўкарыёт, уключала інвазію інтронаў.[60] Вядома пра два канчатковых механізма страты інтронаў: страта інтронаў, апасродкаваная зваротнай транскрыптазай (RTMIL) і геномныя дэлецыі.[61] Канчатковыя механізмы атрымання інтронаў, аднак, застаюцца няўлоўнымі і спрэчнымі. Да гэтага часу паведамлялася як мінімум пра сем механізмаў распаўсюджвання інтронаў: транспазіцыя інтрона, устаўка транспазона, тандэмная геномная дуплікацыя, перанос інтрона, павелічэнне колькасці інтронаў падчас рэпарацыі двухланцуговага разрыву ДНК (далей — DSBR, ад англ. double-strand break repair), устаўка інтрона групы II і інтранізацыя. Тэарэтычна было б прасцей за ўсё вызначыць паходжанне нядаўна атрыманых інтронаў з-за адсутнасці мутацый, выкліканых гаспадаром, але нават нядаўна атрыманыя інтроны ўзніклі шляхам, адрозным ад вышэйзгаданых механізмаў. Такім чынам, гэтыя высновы падымаюць пытанне аб тым, што прапанаваныя механізмы распаўсюджвання інтронаў не могуць апісаць механістычнае паходжанне многіх новых інтронаў, таму што яны не з’яўляюцца дакладнымі механізмамі павелічэння колькасці інтронаў, ці існуюць іншыя працэсы, што спараджаюць новыя інтроны, якія яшчэ трэба будзе адкрыць.[62]
Пры транспазіцыі інтрона, найбольш часта меркаваным механізме распаўсюджання інтронаў, мяркуецца, што сплайсаваны інтрон праходзіць праз зваротны сплайсінг альбо ў сваю ўласную мРНК, альбо ў іншую мРНК у ранейшай пазіцыі без інтрона. Затым гэтая мРНК, што змяшчае інтрон, падвяргаецца зваротнай транскрыпцыі, і атрыманая ў выніку кДНК, з новым інтронам, можа выклікаць распаўсюджванне інтрона шляхам поўнай або частковай рэкамбінацыі са сваім зыходным геномным локусам.
Было паказана, што ўстаўкі транспазонаў ствараюць тысячы новых інтронаў у розных эўкарыятычных відаў.[63] Інсерцыі транспазона часам прыводзяць да дубліравання гэтай паслядоўнасці з кожнага боку транспазона. Такая інсерцыя магла б інтранізаваць транспазон без парушэння кадуючай паслядоўнасці, калі транспазон устаўляецца ў паслядоўнасць AGGT або кадуе сайты сплайсінгу ў паслядоўнасці транспазона. Там, дзе транспазоны, якія генеруюць інтроны, не ствараюць дублікацый сайтаў-мішэняў, элементы ўключаюць абодва сайты сплайсінгу GT (5') і AG (3'), такім чынам, сплайсінг ідзе дакладна, не закранаючы паслядоўнасць, што кадуе бялок.[63] Пакуль незразумела, чаму гэтыя элементы сплайсуюцца, выпадкова ці ў выніку нейкага пераважнага дзеяння транспазона.
У тандэмнай геномнай дуплікацыі з-за падабенства паміж кансэнсуснымі донарскімі і акцэптарнымі сайтамі сплайсінгу, якія абодва вельмі нагадваюць AGGT, тандэмная геномная дуплікацыя экзанічнага сегмента, які змяшчае паслядоўнасць AGGT, стварае два патэнцыйныя сайты сплайсінгу. Пры распазнанні сплайсасомай паслядоўнасць паміж арыгінальным і дубліраваным AGGT будзе сплайсавана, што прывядзе да стварэння інтрона без змены кадуючай паслядоўнасці гена. Рэпарацыя двухланцуговага разрыву праз негамалагічнае злучэнне канцоў нядаўна была ідэнтыфікаваная як крыніца распаўсюджвання інтронаў, калі даследчыкі вызначылі кароткія прамыя паўторы, якія фланкіруюць 43 % атрыманых інтронаў у дафній.[62] Аднак для статыстычнай значнасці, гэтыя лічбы трэба параўноўваць з колькасцю акружаных паўторамі кансерватыўных інтронаў сярод іншых арганізмаў.
Была выказана гіпотэза, што перанос інтрона прыводзіць да павелічэння колькасці інтронаў, калі паралаг або псеўдаген атрымлівае інтрон, а затым пераносіць гэты інтрон шляхам рэкамбінацыі ў месца ў яго роднасным паралагу, дзе няма інтрона. Інтранізацыя — гэта працэс, пры якім мутацыі ствараюць новыя інтроны з ранейшай паслядоўнасці экзона. Такім чынам, у адрозненне ад іншых прапанаваных механізмаў распаўсюджвання інтронаў, гэты механізм не патрабуе інсерцыі або генерацыі ДНК для стварэння новага інтрона.[62]
Адзіны гіпатэтычны механізм распаўсюджвання інтронаў, які не мае ніякіх прамых доказаў, — гэта ўстаўка інтронаў групы II, якая in vivo прыпыняе экспрэсію генаў.[64] Такім чынам, інтроны групы II, хутчэй за ўсё, з’яўляюцца меркаванымі продкамі сплайсасомных інтронаў, якія дзейнічаюць як сайт-спецыфічныя рэтраэлементы і больш не адказваюць за распаўсюджванне інтронаў.[65][66] Тандэмная геномная дуплікацыя — адзіны прапанаваны механізм, які пацвярджаюць эксперыментальныя даныя in vivo: кароткая ўнутрыгенная тандэмная дуплікацыя можа ўставіць новы інтрон у ген, які кадуе бялок, пакідаючы адпаведную паслядоўнасць пептыда нязменнай.[67] Гэты механізм таксама мае шырокія ўскосныя доказы, якія пацвярджаюць ідэю, што тандэмная геномная дуплікацыя з’яўляецца распаўсюджаным механізмам павелічэння колькасці інтронаў. Тэставанне іншых прапанаваных механізмаў in vivo, у прыватнасці распаўсюджвання інтронаў праз DSBR, транспарт інтронаў і інтранізацыі, магчыма, хоць гэтыя механізмы павінны быць прадэманстраваны in vivo, каб замацаваць іх як фактычныя механізмы атрымання інтронаў генамі. Далейшы геномны аналіз, асабліва на папуляцыйным узроўні, можа колькасна вызначыць адносны ўклад кожнага механізму, магчыма, выяўляючы відаспецыфічныя перавагі, якія могуць праліць святло на розную хуткасць распаўсюджвання інтронаў сярод розных відаў.[62]
Гл. таксама
[правіць | правіць зыходнік]Структура:
Сплайсінг:
Функцыя
Іншае:
Крыніцы
[правіць | правіць зыходнік]- ↑ а б «The notion of the cistron [i.e., gene] … must be replaced by that of a transcription unit containing regions which will be lost from the mature messenger — which I suggest we call introns (for intragenic regions) — alternating with regions which will be expressed — exons.» (Gilbert 1978) Gilbert W (February 1978). "Why genes in pieces?". Nature. 271 (5645): 501. Bibcode:1978Natur.271..501G. doi:10.1038/271501a0. PMID 622185. S2CID 4216649.
- ↑ Kinniburgh AJ, Mertz JE, Ross J (July 1978). "The precursor of mouse beta-globin messenger RNA contains two intervening RNA sequences". Cell. 14 (3): 681–693. doi:10.1016/0092-8674(78)90251-9. PMID 688388. S2CID 21897383.
- ↑ Lewin B (1987). Genes (3rd ed.). New York: Wiley. pp. 159–179, 386. ISBN 0-471-83278-2. OCLC 14069165.
- ↑ Chow LT, Gelinas RE, Broker TR, Roberts RJ (September 1977). "An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA". Cell. 12 (1): 1–8. doi:10.1016/0092-8674(77)90180-5. PMID 902310. S2CID 2099968.
- ↑ Berget SM, Moore C, Sharp PA (August 1977). "Spliced segments at the 5' terminus of adenovirus 2 late mRNA". Proceedings of the National Academy of Sciences of the United States of America. 74 (8): 3171–3175. Bibcode:1977PNAS...74.3171B. doi:10.1073/pnas.74.8.3171. PMC 431482. PMID 269380.
- ↑ Belfort M, Pedersen-Lane J, West D, Ehrenman K, Maley G, Chu F, Maley F (June 1985). "Processing of the intron-containing thymidylate synthase (td) gene of phage T4 is at the RNA level". Cell. 41 (2): 375–382. doi:10.1016/s0092-8674(85)80010-6. PMID 3986907. S2CID 27127017.
- ↑ The Nobel Prize in Physiology or Medicine 1993.
- ↑ Tonegawa S, Maxam AM, Tizard R, Bernard O, Gilbert W (March 1978). "Sequence of a mouse germ-line gene for a variable region of an immunoglobulin light chain". Proceedings of the National Academy of Sciences of the United States of America. 75 (3): 1485–1489. Bibcode:1978PNAS...75.1485T. doi:10.1073/pnas.75.3.1485. PMC 411497. PMID 418414.
- ↑ Tilghman SM, Tiemeier DC, Seidman JG, Peterlin BM, Sullivan M, Maizel JV, Leder P (February 1978). "Intervening sequence of DNA identified in the structural portion of a mouse beta-globin gene". Proceedings of the National Academy of Sciences of the United States of America. 75 (2): 725–729. Bibcode:1978PNAS...75..725T. doi:10.1073/pnas.75.2.725. PMC 411329. PMID 273235.
- ↑ Stajich JE, Dietrich FS, Roy SW (2007). "Comparative genomic analysis of fungal genomes reveals intron-rich ancestors". Genome Biology. 8 (10): R223. doi:10.1186/gb-2007-8-10-r223. PMC 2246297. PMID 17949488.
- ↑ Taanman JW (February 1999). "The mitochondrial genome: structure, transcription, translation and replication". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1410 (2): 103–123. doi:10.1016/s0005-2728(98)00161-3. PMID 10076021. S2CID 19229072.
- ↑ Tollervey D, Caceres JF (November 2000). "RNA processing marches on". Cell. 103 (5): 703–709. doi:10.1016/S0092-8674(00)00174-4. PMID 11114327.
- ↑ Reugels AM, Kurek R, Lammermann U, Bünemann H (February 2000). "Mega-introns in the dynein gene DhDhc7(Y) on the heterochromatic Y chromosome give rise to the giant threads loops in primary spermatocytes of Drosophila hydei". Genetics. 154 (2): 759–769. doi:10.1093/genetics/154.2.759. PMC 1460963. PMID 10655227.
- ↑ Piovesan A, Caracausi M, Ricci M, Strippoli P, Vitale L, Pelleri MC (December 2015). "Identification of minimal eukaryotic introns through GeneBase, a user-friendly tool for parsing the NCBI Gene databank". DNA Research. 22 (6): 495–503. doi:10.1093/dnares/dsv028. PMC 4675715. PMID 26581719.
- ↑ Slabodnick MM, Ruby JG, Reiff SB, Swart EC, Gosai S, Prabakaran S, et al. (February 2017). "The Macronuclear Genome of Stentor coeruleus Reveals Tiny Introns in a Giant Cell". Current Biology. 27 (4): 569–575. doi:10.1016/j.cub.2016.12.057. PMC 5659724. PMID 28190732.
- ↑ Copertino DW, Hallick RB (December 1993). "Group II and group III introns of twintrons: potential relationships with nuclear pre-mRNA introns". Trends in Biochemical Sciences. 18 (12): 467–471. doi:10.1016/0968-0004(93)90008-b. PMID 8108859.
- ↑ Padgett RA, Grabowski PJ, Konarska MM, Seiler S, Sharp PA (1986). "Splicing of messenger RNA precursors". Annual Review of Biochemistry. 55: 1119–1150. doi:10.1146/annurev.bi.55.070186.005351. PMID 2943217.
- ↑ Guthrie C, Patterson B (1988). "Spliceosomal snRNAs". Annual Review of Genetics. 22: 387–419. doi:10.1146/annurev.ge.22.120188.002131. PMID 2977088.
- ↑ Greer CL, Peebles CL, Gegenheimer P, Abelson J (February 1983). "Mechanism of action of a yeast RNA ligase in tRNA splicing". Cell. 32 (2): 537–546. doi:10.1016/0092-8674(83)90473-7. PMID 6297798. S2CID 44978152.
- ↑ Reinhold-Hurek B, Shub DA (May 1992). "Self-splicing introns in tRNA genes of widely divergent bacteria". Nature. 357 (6374): 173–176. Bibcode:1992Natur.357..173R. doi:10.1038/357173a0. PMID 1579169. S2CID 4370160.
- ↑ Cech TR (1990). "Self-splicing of group I introns". Annual Review of Biochemistry. 59: 543–568. doi:10.1146/annurev.bi.59.070190.002551. PMID 2197983.
- ↑ Michel F, Ferat JL (1995). "Structure and activities of group II introns". Annual Review of Biochemistry. 64: 435–461. doi:10.1146/annurev.bi.64.070195.002251. PMID 7574489.
- ↑ Wan R, Bai R, Zhan X, Shi Y (2020). "How is precursor messenger RNA spliced by the spliceosome?". Annual Review of Biochemistry. 89: 333–358. doi:10.1146/annurev-biochem-013118-111024. PMID 31815536. S2CID 209167227.
- ↑ Wilkinson ME, Charenton C, Nagai K (2020). "RNA splicing by the spliceosome". Annual Review of Biochemistry. 89: 359–388. doi:10.1146/annurev-biochem-091719-064225. PMID 31794245. S2CID 208626110.
- ↑ Sales-Lee J, Perry DS, Bowser BA, Diedrich JK, Rao B, Beusch I, Yates III JR, Roy SW, Madhani HD (2021). "Coupling of spliceosome complexity to intron diversity". Current Biology. 31 (22): 4898–4910 e4894. doi:10.1016/j.cub.2021.09.004. PMC 8967684. PMID 34555349. S2CID 237603074.
- ↑ Hsu SN, Hertel KJ (2009). "Spliceosomes walk the line: splicing errors and their impact on cellular function". RNA Biology. 6 (5): 526–530. doi:10.4161/rna.6.5.9860. PMC 3912188. PMID 19829058. S2CID 22592978.
- ↑ Melamud E, Moult J (2009). "Stochastic noise in splicing machinery". Nucleic Acids Research. gkp471 (14): 4873–4886. doi:10.1093/nar/gkp471. PMC 2724286. PMID 19546110.
- ↑ а б Fox-Walsh KL, Hertel KJ (2009). "Splice-site pairing is an intrinsically high fidelity process". Proceedings of the National Academy of Sciences. 106 (6): 1766–1771. Bibcode:2009PNAS..106.1766F. doi:10.1073/pnas.0813128106. PMC 2644112. PMID 19179398.
- ↑ Stepankiw N, Raghavan M, Fogarty EA, Grimson A, Pleiss JA (2015). "Widespread alternative and aberrant splicing revealed by lariat sequencing". Nucleic Acids Research. 43 (17): 8488–8501. doi:10.1093/nar/gkv763. PMC 4787815. PMID 26261211.
- ↑ а б в Pickrell JK, Pai AA, Gilad Y, Pritchard JK (2010). "Noisy splicing drives mRNA isoform diversity in human cells". PLOS Genet. 6 (12): e1001236. doi:10.1371/journal.pgen.1001236. PMC 3000347. PMID 21151575.
- ↑ а б Skandalis A (2016). "Estimation of the minimum mRNA splicing error rate in vertebrates". Mutation Research/Fundamental and Molecular Mechanisms of Mutagenesis. 784 (1713): 34–38. doi:10.1098/rstb.2015.0474. PMC 5182408. PMID 27994117.
- ↑ Zhang Z, Xin D, Wang P, Zhou L, Hu L, Kong X, Hurst LD (2009). "Noisy splicing, more than expression regulation, explains why some exons are subject to nonsense-mediated mRNA decay". BMC Biology. 7: 23. doi:10.1186/1741-7007-7-23. PMC 2697156. PMID 19442261.
- ↑ Bitton DA, Atkinson SR, Rallis C, Smith GC, Ellis DA, Chen YY, Malecki M, Codlin S, Lemay JF, Cotobal C (2015). "Widespread exon skipping triggers degradation by nuclear RNA surveillance in fission yeast". Genome Research. 25 (6): 884–896. doi:10.1101/gr.185371.114. PMC 4448684. PMID 25883323.
- ↑ Saudemont B, Popa A, Parmley JL, Rocher V, Blugeon C, Necsulea A, Meyer E, Duret L (2017). "The fitness cost of mis-splicing is the main determinant of alternative splicing patterns". Genome Biology. 18 (1): 208. doi:10.1186/s13059-017-1344-6. PMC 5663052. PMID 29084568.
- ↑ Scotti MM, Swanson MS (2016). "RNA mis-splicing in disease". Nature Reviews Genetics. 17 (1): 19–32. doi:10.1038/nrg.2015.3. PMC 5993438. PMID 26593421.
- ↑ а б Shirley B, Mucaki E, Rogan P (2019). "Pan-cancer repository of validated natural and cryptic mRNA splicing mutations". F1000Research. 7: 1908. doi:10.12688/f1000research.17204.3. PMC 6544075. PMID 31275557. S2CID 202702147.
- ↑ Mucaki EJ, Shirley BC, Rogan PK (2020). "Expression changes confirm genomic variants predicted to result in allele-specific, alternative mRNA splicing". Frontiers in Genetics. 11: 109. doi:10.3389/fgene.2020.00109. PMC 7066660. PMID 32211018.
- ↑ Rogaev EI, Grigorenko AP, Faskhutdinova G, Kittler EL, Moliaka YK (2009). "Genotype analysis identifies the cause of the "royal disease"". Science. 326 (5954): 817. Bibcode:2009Sci...326..817R. doi:10.1126/science.1180660. PMID 19815722. S2CID 206522975.
- ↑ Lynch M (2010). "Rate, molecular spectrum, and consequences of human mutation". Proceedings of the National Academy of Sciences. 107 (3): 961–968. Bibcode:2010PNAS..107..961L. doi:10.1073/pnas.0912629107. PMC 2824313. PMID 20080596.
- ↑ Mudge JM, Harrow J (2016). "The state of play in higher eukaryote gene annotation". Nature Reviews Genetics. 17 (12): 758–772. doi:10.1038/nrg.2016.119. PMC 5876476. PMID 27773922.
- ↑ Bhuiyan SA, Ly S, Phan M, Huntington B, Hogan E, Liu CC, Liu J, Pavlidis P (2018). "Systematic evaluation of isoform function in literature reports of alternative splicing". BMC Genomics. 19 (1): 637. doi:10.1186/s12864-018-5013-2. PMC 6114036. PMID 30153812.
- ↑ Rearick D, Prakash A, McSweeny A, Shepard SS, Fedorova L, Fedorov A (March 2011). "Critical association of ncRNA with introns". Nucleic Acids Research. 39 (6): 2357–2366. doi:10.1093/nar/gkq1080. PMC 3064772. PMID 21071396.
- ↑ Bicknell AA, Cenik C, Chua HN, Roth FP, Moore MJ (December 2012). "Introns in UTRs: why we should stop ignoring them". BioEssays. 34 (12): 1025–1034. doi:10.1002/bies.201200073. PMID 23108796. S2CID 5808466.
- ↑ Cenik C, Chua HN, Zhang H, Tarnawsky SP, Akef A, Derti A, et al. (April 2011). Snyder M (рэд.). "Genome analysis reveals interplay between 5'UTR introns and nuclear mRNA export for secretory and mitochondrial genes". PLOS Genetics. 7 (4): e1001366. doi:10.1371/journal.pgen.1001366. PMC 3077370. PMID 21533221.
- ↑ Penny D, Hoeppner MP, Poole AM, Jeffares DC (November 2009). "An overview of the introns-first theory". Journal of Molecular Evolution. 69 (5): 527–540. Bibcode:2009JMolE..69..527P. doi:10.1007/s00239-009-9279-5. PMID 19777149. S2CID 22386774.
- ↑ Cavalier-Smith T (1991). "Intron phylogeny: a new hypothesis". Trends in Genetics. 7 (5): 145–148. doi:10.1016/0168-9525(91)90377-3. PMID 2068786.
- ↑ Doolittle WF (1991). "The origins of introns". Current Biology. 1 (3): 145–146. doi:10.1016/0960-9822(91)90214-h. PMID 15336149. S2CID 35790897.
- ↑ Sharp PA (1991). ""Five easy pieces."(role of RNA catalysis in cellular processes)". Science. 254 (5032): 663–664. doi:10.1126/science.1948046. PMID 1948046. S2CID 508870.
- ↑ Irimia M, and Roy SW (2014). "Origin of spliceosomal introns and alternative splicing". Cold Spring Harbor Perspectives in Biology. 6 (6): a016071. doi:10.1101/cshperspect.a016071. PMC 4031966. PMID 24890509.
- ↑ Rodríguez-Trelles F, Tarrío R, Ayala FJ (2006). "Origins and evolution of spliceosomal introns". Annual Review of Genetics. 40: 47–76. doi:10.1146/annurev.genet.40.110405.090625. PMID 17094737.
- ↑ Mourier T, Jeffares DC (May 2003). "Eukaryotic intron loss". Science. 300 (5624): 1393. doi:10.1126/science.1080559. PMID 12775832. S2CID 7235937.
- ↑ Roy SW, Gilbert W (March 2006). "The evolution of spliceosomal introns: patterns, puzzles and progress". Nature Reviews. Genetics. 7 (3): 211–221. doi:10.1038/nrg1807. PMID 16485020. S2CID 33672491.
- ↑ de Souza SJ (July 2003). "The emergence of a synthetic theory of intron evolution". Genetica. 118 (2–3): 117–121. doi:10.1023/A:1024193323397. PMID 12868602. S2CID 7539892.
- ↑ Lynch M (April 2002). "Intron evolution as a population-genetic process". Proceedings of the National Academy of Sciences of the United States of America. 99 (9): 6118–6123. Bibcode:2002PNAS...99.6118L. doi:10.1073/pnas.092595699. PMC 122912. PMID 11983904.
- ↑ Jeffares DC, Mourier T, Penny D (January 2006). "The biology of intron gain and loss". Trends in Genetics. 22 (1): 16–22. doi:10.1016/j.tig.2005.10.006. PMID 16290250.
- ↑ Jeffares DC, Penkett CJ, Bähler J (August 2008). "Rapidly regulated genes are intron poor". Trends in Genetics. 24 (8): 375–378. doi:10.1016/j.tig.2008.05.006. PMID 18586348.
- ↑ Castillo-Davis CI, Mekhedov SL, Hartl DL, Koonin EV, Kondrashov FA (August 2002). "Selection for short introns in highly expressed genes". Nature Genetics. 31 (4): 415–418. doi:10.1038/ng940. PMID 12134150. S2CID 9057609.
- ↑ а б в Bonnet A, Grosso AR, Elkaoutari A, Coleno E, Presle A, Sridhara SC, et al. (August 2017). "Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability". Molecular Cell. 67 (4): 608–621.e6. doi:10.1016/j.molcel.2017.07.002. PMID 28757210.
- ↑ Parenteau J, Maignon L, Berthoumieux M, Catala M, Gagnon V, Abou Elela S (January 2019). "Introns are mediators of cell response to starvation". Nature. 565 (7741): 612–617. Bibcode:2019Natur.565..612P. doi:10.1038/s41586-018-0859-7. PMID 30651641. S2CID 58014466.
- ↑ Rogozin IB, Carmel L, Csuros M, Koonin EV (April 2012). "Origin and evolution of spliceosomal introns". Biology Direct. 7: 11. doi:10.1186/1745-6150-7-11. PMC 3488318. PMID 22507701.
- ↑ Derr LK, Strathern JN (January 1993). "A role for reverse transcripts in gene conversion". Nature. 361 (6408): 170–173. Bibcode:1993Natur.361..170D. doi:10.1038/361170a0. PMID 8380627. S2CID 4364102.
- ↑ а б в г Yenerall P, Zhou L (September 2012). "Identifying the mechanisms of intron gain: progress and trends". Biology Direct. 7: 29. doi:10.1186/1745-6150-7-29. PMC 3443670. PMID 22963364.
- ↑ а б Gozashti L, Roy S, Thornlow B, Kramer A, Ares M, Corbett-Detig R (November 2022). "Transposable elements drive intron gain in diverse eukaryotes". PNAS. 119 (48): 48. doi:10.1073/pnas.2209766119. PMC 9860276. PMID 36417430.
- ↑ Chalamcharla VR, Curcio MJ, Belfort M (April 2010). "Nuclear expression of a group II intron is consistent with spliceosomal intron ancestry". Genes & Development. 24 (8): 827–836. doi:10.1101/gad.1905010. PMC 2854396. PMID 20351053.
- ↑ Cech TR (January 1986). "The generality of self-splicing RNA: relationship to nuclear mRNA splicing". Cell. 44 (2): 207–210. doi:10.1016/0092-8674(86)90751-8. PMID 2417724. S2CID 11652546.
- ↑ Dickson L, Huang HR, Liu L, Matsuura M, Lambowitz AM, Perlman PS (November 2001). "Retrotransposition of a yeast group II intron occurs by reverse splicing directly into ectopic DNA sites". Proceedings of the National Academy of Sciences of the United States of America. 98 (23): 13207–13212. Bibcode:2001PNAS...9813207D. doi:10.1073/pnas.231494498. PMC 60849. PMID 11687644.
- ↑ Hellsten U, Aspden JL, Rio DC, Rokhsar DS (August 2011). "A segmental genomic duplication generates a functional intron". Nature Communications. 2: 454. Bibcode:2011NatCo...2..454H. doi:10.1038/ncomms1461. PMC 3265369. PMID 21878908.
Знешнія спасылкі
[правіць | правіць зыходнік]- A search engine for exon/intron sequences defined by NCBI
- Bruce Alberts, Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walter Molecular Biology of the Cell, 2007, ISBN 978-0-8153-4105-5. Fourth edition is available online through the NCBI Bookshelf: link
- Jeremy M Berg, John L Tymoczko, and Lubert Stryer, Biochemistry 5th edition, 2002, W H Freeman. Available online through the NCBI Bookshelf: link
- Intron finding tool for plant genomic sequences
- Exon-intron graphic maker
